George Church:多重基因组工程与测试(二)
发布时间:2022-11-24讲 座 主 题
关于多重基因组工程(multiplex genome engineering)以及我们如何用它来进行新应用的测试。
主 讲 人 简 介

George Church
哈佛医学院遗传学教授、美国科学院院士、美国工程院院士、富兰克林鲍尔科学成就奖得主。Church合著超过625篇论文,拥有156项专利和一本著作《Regenesis》。Church在基因组学和合成生物学领域卓有建树,他开发了首个基因组测序方法,大大降低了测序成本,为几乎所有“下一代测序”方法和和公司做出了贡献。此外,他的团队发明了用于人类干细胞和基因组编辑和其他合成生物学应用的CRISPR技术,包括创建器官移植方法、逆转衰老的基因疗法等。
讲 座 内 容
基因编辑的挑战不仅仅在于如何实现精确编辑,更在于如何使它们能真正发挥作用。以下展示的是一些基因编辑通用的递送方法,包括电穿孔、基因枪、脂质体递送、细胞融合、病毒衣壳载体等等。我们也在尝试使用细胞和生殖系做一些实验。
这里我们重点介绍一下病毒载体。对于动物宿主,我们有各种各样的病毒载体,其中一些可能会引起人畜共患病,如艾滋病、天花、脊髓灰质炎等等都是罕见的人畜共患病,此外还有会经常性反复发生的猪流感、禽流感、黄热病毒、寨卡病毒等等。
这反映了这些病毒的组织嗜性(tissue tropism)和宿主趋向性。我们在一个生物体中测试病毒的抗性时发现,其临近物种都对这种病毒非常易感。现在,我们所做的是尝试用一种通用的方法来研究和设计它们。
为了同时进行学习和使用,我们结合了机器学习和巨库技术(megalibrary),通过一种循环迭代的方式【编者注:DNA合成-筛选-动物实验和测序-AI学习-新一轮设计合成】,我们可以对数据库测序,并针对免疫逃逸和组织噬性进行选择。随后,通过机器学习尽可能从数百个样本中心收集数据并迭代。得到结果后,再用来进一步设计蛋白质和基因组。
在我们的样本库中,拥有着数百万,甚至数十亿个不同分子的组合,这使我们可以高效低成本地进行临床试验【编者注:应用于许多不同形式的基因治疗及大规模计算机辅助蛋白质设计和试错迭代】。
我们有四篇相关论文已经发表,其中两篇是关于使用深度学习或机器学习的。这四篇文章随后孵化了四家公司,Nabla Bio专注于酶和抗体,ManifoldBio主要研究蛋白质疗法,Dyno Therapeutics正在设计改良工程化AAV递送,Patch Bioscience则是研究工程化核酸顺式作用元件。

进一步地,我们能够利用这些多重大型文库,通过机器学习制造优化,例如构建具有超过一百万个不同变体的AAV库,每个变体都有不同的衣壳和基因组条形码,可以在体内或体外测试免疫和组织特异性。我们可以将它注射到动物体内,在不同组织中提取样本,并以此来研究感兴趣的组织中的最佳递送方法或设法规避此类递送方法。
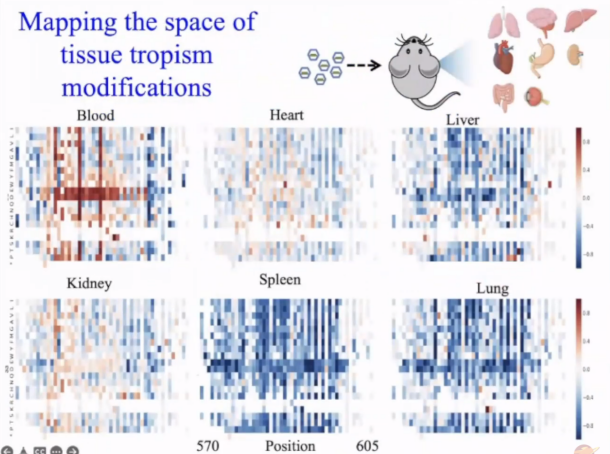
Pierce和Eric在Science发表了一篇相关论文,并基于此成立了Dyno Therapeutics。图中是我们采集的六个组织中的基本数据热图,X轴是递送胶囊(capsule)相关改造在基因组中的位置,Y轴是密码子(插入及替换)。
其中的红色意味着在特定组织中存在有利突变【编者注:白色代表没有变化,蓝色为不利突变】,每一个点,每一个矩形代表一个独立的突变(本质上是一个独立的病毒被设计和读出的结果)。
所以,通过研究不同突变在动物模型内的表现,研究人员可以发现某些突变非常富集的组织,比如心脏。也可以发现突变在体内不同的趋向性或归巢性。
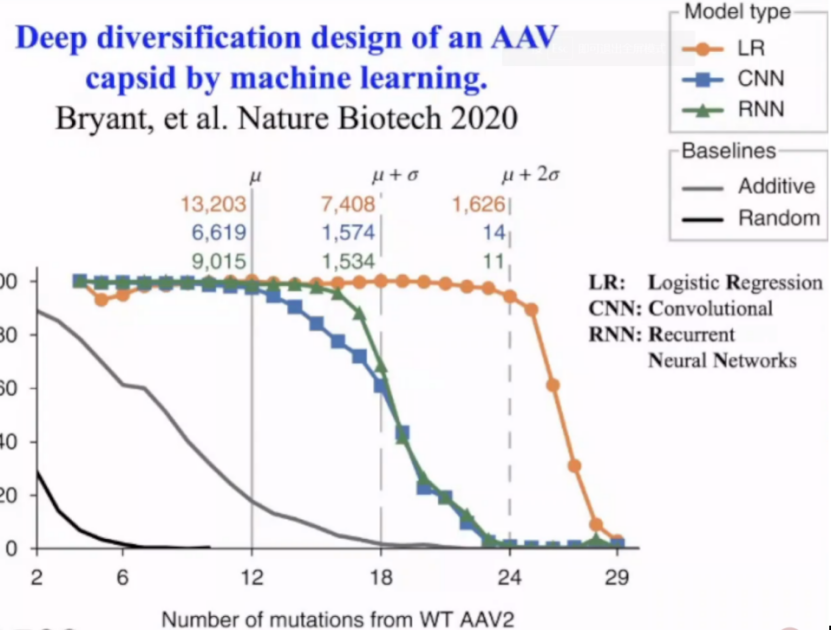
【编者注:工程蛋白文库的序列多样性很少超过天然蛋白家族】在基因组的每个位置引入编辑,探究我们感兴趣的蛋白质功能。下面这个例子是基于深度学习的AAV衣壳高度多样化设计,于2020年发表在Nature Biotech。其中,X轴是突变数,Y轴是生存能力。
即使训练的数据有限,深度神经网络模型也能准确地预测衣壳突变如何影响不同变体的生存能力。【编者注:这种方法打开了大量具有功能性但曾经无法到达的序列空间,在病毒载体和蛋白质疗法等领域有许多潜在应用】
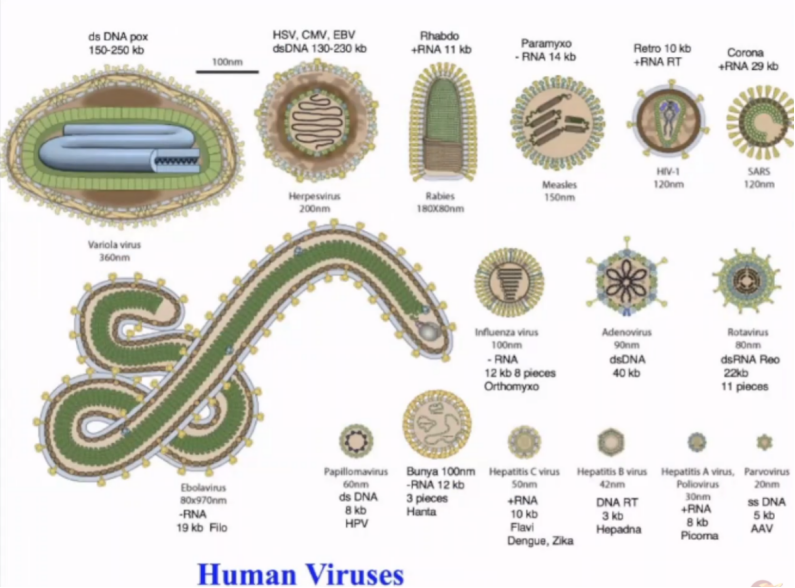
下面这张图展示了一些能够感染人类的病毒。其中有我们之前已经提到的,比如图中左上角的疱疹病毒,以及右下方的AAV等病毒,我们通常用它们来构建载体。
另外,还有一些病原体,像冠状病毒。这些病毒可以进入安全等级很高的无菌环境,偶尔也可能通过培养基传播。比如水疱疹病毒vesivirus,它影响了欧洲和美国的工厂长达两年之久(值得注意的是,这种情况发生在工厂里,不是在野外。)
所以可以想象,病毒对于微生物、植物、动物、人类、甚至工业环境都可能造成很大的问题。当然,我们也有很多相应方法能够对抗它们。你可以一次对付一类【编者注:病毒特异性受体】,就像CCR5对HIV,但这个受体最好不重要或者不会伤害正常细胞。实际上,这一点很难做到,所以这种方法也难以推广。
有些病毒特异性的核酸酶(比如限制性内切酶)和CRISPR,它们在细菌中很有效,但据我所知相关产品尚未进入临床试验。而对于人类而言,我们已经开发出内源性拷贝(endogenous copies)的方法,比如使用内源性逆转录病毒(Endogenous Retrpvirus, ERV)【编者注:ERV是基因组转座子元件之一】,稍后还会提到。
我们也研究过全基因组重编码,【编者注:George Church与杨璐菡合作,使用全基因组重编程技术清除了猪基因组中的内源性逆转录病毒基因,相关成果发表于Science】。
以上提到的这些对于人类来说都是非常令人兴奋的应用,但在这里我们先仅就工业微生物进行讨论,因为我们认为这已经足以阐明其基本原理,且它普遍适用于任何生物,包括对所有自然病毒,甚至是我们从未见过的病毒产生抗性。
近期,我们对野生型以及和新分离的病毒进行了测试。一般情况下,我们能做的都仅是改变64个三联密码子中的一个或多个。在最初的研究中,我们仅仅是改变了最简单的一个密码子【编者注:UAG-UAA】,对应改变了321个基因(2013年5月)。而在我们最新重编码的基因组中,则包含了高达62214个密码子位点的改变(2016年9月)。
期间,我们还进行过一些其他项目,例如我们通过改变更多密码子,使任意生物能够对所有病毒产生抗性。这个项目从2009年发表第一篇文章开始,直到现在仍在继续进行,最新的研究工作有望在明年发表。

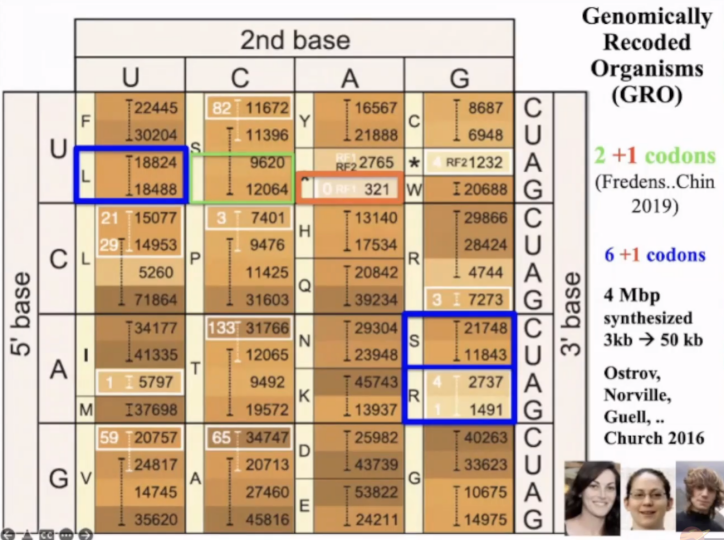
在这个遗传密码表中,小字表示密码子在大肠杆菌基因组中的发生次数,框选的是我们已经实现的重编码目标。
红色框是我们做的第一个密码子重编码,发表在2013年,我们将其改成一个终止密码子。绿色框是Jason Chin实验室的工作。我们在基本相同的底盘菌株中,研究了蓝色框中的另外6个密码子,并创造了基因组重编码有机体(genomically recorded organisms, GRO)。
重编码后的生物可以抵抗多种病毒,我们实验室在13年发表了相关论文,Isaacs和Chin也分别在16年和21年发表了有关成果。
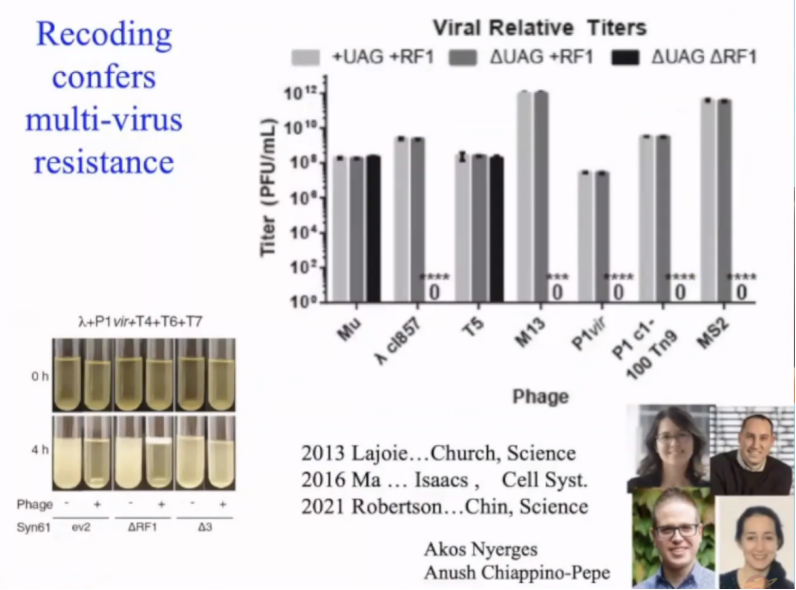
我们还是希望尽快发表一篇论文证明新编码的生物体可以对所有病毒都表现出抗性。如图所示,【编者注:只改变了UAG】你可以对应不同病毒的数据中中看到“0”【编者注:六种病毒中的四种被成功拦截】,只有Mu和T5是两个例外。
这是一个非常戏剧性的结果,你可以理解为,每毫升一千亿个(1011个)同种病毒仅仅因为一个简单的改变,其数量就被压制为零(无法增殖)。据我所知,这些工作还没有在最近的多重耐药菌株中进行测试,但看起来是很有希望的,这部分工作正由Akos和Anush进行。
-----
作者/疯与鸽
初审/短腿小马达
复审/捉蝴蝶的猫
编辑/果粒珍珍


